基于全基因组测序的耐碳青霉烯类抗生素鲍曼不动杆菌耐药基因、毒力因子及同源性分析

打开文本图片集
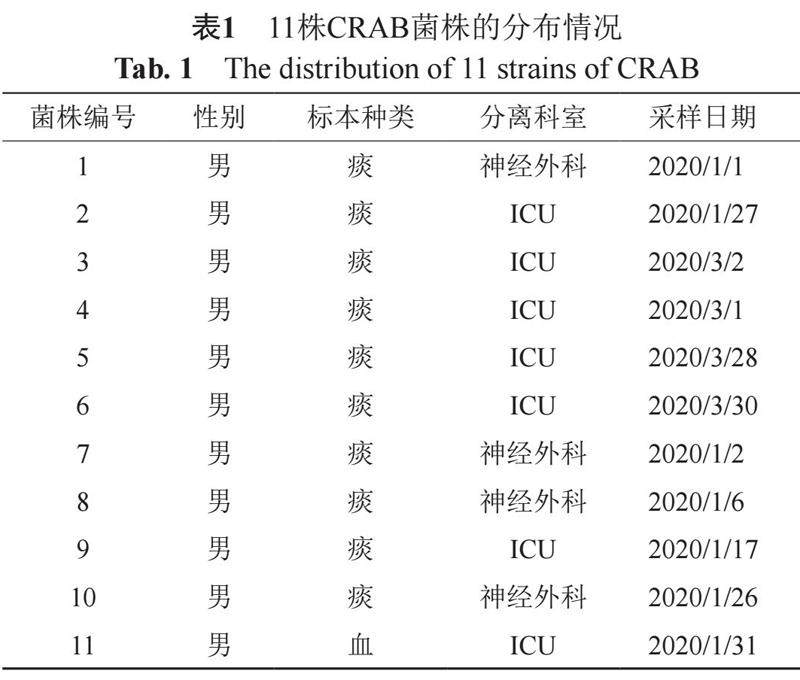
摘要:目的 通过应用全基因组测序(whole genome sequencing, WGS)技术分析某三级医疗机构耐碳青霉烯类抗生素鲍曼不动杆菌(carbapenem-resistant Acinetobacter baumannii, CRAB)的耐药基因、毒力因子及同源性。方法 收集该院2020年1月至3月重症监护病房(Intensive care unit, ICU)、神经外科分离的11株医院感染CRAB菌株,通过二代测序平台进行全基因组测序, 应用基因组流行病学中心(Center for genomic epidemiology, CGE)ResFinder 4. 0软件分析其耐药基因型,并应用MORPHEUS在线制作热图,应用毒力因子数据库(virulence factors of pathogenic bacteria, VFDB)VFanalyzer软件筛选毒力因子,应用MLST软件检测菌株的ST型,应用Kaptive软件检测荚膜型,应用CSI Phylogeny 1. 4软件及FigTree v1. 4. 4软件构建最大似然树(maximum likelihood tree, MLT)以分析其同源性。(剩余16231字)